Forschende entdecken gemeinsame Evolutionsgeschichte von Darmmikroben und ihren menschlichen Wirten
Überschriften einblenden ≡
Viele Mikroben-Spezies im menschlichen Darm sind in verschiedensten Bevölkerungsgruppen auf der ganzen Welt zu finden. Die Mikroben-Stämme innerhalb einer Mikroben-Art unterscheiden sich jedoch erheblich zwischen Individuen und Populationen. Trotz ihrer Bedeutung für die menschliche Gesundheit war bisher wenig über die Entwicklung der verschiedenen Stämme bekannt. Angesichts dessen, dass zudem die meisten dieser Stämme fast ausschließlich im menschlichen Darm vorkommen, stellt sich die Frage nach ihrer Herkunft.
Das Forschungsteam des Max-Planck-Instituts für Biologie in Tübingen stellte die Vermutung auf, dass bestimmte Arten und Stämme den Menschen bei der Ausbreitung über die Erde begleitet und sich in seinem Darm parallel mitentwickelt haben. Dazu verglichen die Forschenden des Max-Planck-Instituts für Biologie, des Instituts für Tropenmedizin und des Exzellenzclusters CMFI der Universität Tübingen erstmals systematisch die Evolutionsgeschichten von Menschen und Darmmikroben. Die Forschenden erstellten dazu Stammbäume für 1225 menschliche Versuchspersonen sowie für 59 Mikroben-Arten aus deren Darm. Mithilfe von statistischen Tests analysierten sie, wie gut diese Stammbäume übereinstimmten.
Über 60 Prozent der untersuchten Spezies zeigten eine parallele Stammesgeschichte zu ihrem menschlichen Wirt. Dies weist darauf hin, dass sich diese Mikroben über Hunderttausende Jahre hinweg im menschlichen Darm weiterentwickelten, während die Menschen sich von Afrika aus über die Kontinente ausbreiteten. „Wir wussten bisher nicht, dass unsere Darmmikroben unserer Evolutionsgeschichte so genau gefolgt sind“, staunt Ruth Ley, die Leiterin der Abteilung für Mikrobiomforschung am Max-Planck-Institut für Biologie Tübingen, wo die Studie durchgeführt wurde, sowie stellvertretende Sprecherin des CMFI ist.
Darmmikroben abhängig vom Menschen
„Bemerkenswert ist auch, dass diejenigen Stämme, die unserer Entwicklungsgeschichte am engsten gefolgt sind, nun am meisten von der Darmumgebung abhängig sind“, fügt Ley hinzu. Tatsächlich sind einige der Mikroben-Stämme, die sich zusammen mit dem Menschen entwickelt haben, stark von der menschlichen Darmumgebung abhängig: Sie besitzen kleinere Genome und reagieren empfindlicher auf Abweichungen in Sauerstoffgehalt und Temperatur – Merkmale, die ein Überleben außerhalb des menschlichen Körpers erschweren.
Im Gegensatz dazu wiesen Mikroorganismen, die eine schwächere Verbindung mit der menschlichen Geschichte aufwiesen, mehr Eigenschaften von freilebenden Bakterien auf. „Einige der Darmmikroben verhalten sich, als seien sie Teil des menschlichen Erbguts“, erklärt Taichi Suzuki, der zusammen mit seinem Kollegen Liam Fitzstevens Erstautor der Studie ist. Suzuki ergänzt: „Diese Mikroben befinden sich sozusagen irgendwo im Spektrum von ‘freilebend’ bis hin zu abhängig von der menschlichen Körperumgebung. Wir konnten zeigen, dass einige Darmbakterien des Menschen in diesem Spektrum weiter in Richtung irreversibler Abhängigkeit gekommen sind als bisher angenommen.“ Ley fügt weiter hinzu: „Diese Ergebnisse verändern unsere Sichtweise auf das menschliche Darmmikrobiom grundlegend.“
Auf Bevölkerungsgruppen angepasste Mikrobiom-Therapien
Um Daten einer breitgefächerten Stichprobe unterschiedlicher Bevölkerungsgruppen weltweit zu erhalten, analysierte das Forschungsteam die Darmmikroben und Genome von 1225 Personen in Europa, Asien und Afrika. Die Stuhl- und Speichelproben wurden mit Hilfe von Forschenden des Instituts für Tropenmedizin der Universität Tübingen und deren Partnerorganisationen in Vietnam und Gabun gesammelt. Darüber hinaus unterstützten Forschende auf der ganzen Welt die Studie mit vergleichbaren Datensätze von Teilnehmenden aus Kamerun, Südkorea und dem Vereinigten Königreich.
Die Ergebnisse der Studie tragen zum besseren Verständnis von Mikroben bei, die seit langem zu bestimmten Bevölkerungsgruppen gehören. Mikrobiom-Krankheitstherapien können mit diesem Wissen besser auf die jeweilige lokale Bevölkerung angepasst und verfeinert werden.
Originalveröffentlichung
Taichi A. Suzuki, J. Liam Fitzstevens, Victor T. Schmidt, Hagay Enav, Kelsey E. Huus, Mirabeau Mbong Ngwese, Anne Grießhammer, Anne Pfleiderer, Bayode R. Adegbite, Jeannot F. Zinsou, Meral Esen, Thirumalaisamy P. Velavan, Ayola A. Adegnika, Le Huu Song, Timothy D. Spector, Amanda L. Muehlbauer, Nina Marchi, Hyena Kang, Lisa Maier, Ran Blekhman, Laure Ségurel, GwangPyo Ko, Nicholas D. Youngblut, Peter Kremsner, Ruth E. Ley
Codiversification of gut microbiota with humans.
Science, Vol. 377, Issue 6612 (2022)
https://dx.doi.org/10.1126/science.abm7759
Info-Krümel
Materialien zur Verfügung gestellt durch: Max-Planck-Gesellschaft
Inhalt kann in Stil und Länge editiert sein.
Journal-Referenz:
https://www.mpg.de/19220836/0915-entw-gemeinsame-evolution-darmmikroben-und-ihre-menschen-151730-x
Tooltips:
Die Texte in eventuell vorhandenen Tooltip-Fenstern wurden von der Redaktion des W3punkt.de bereitgestellt, sie entstammen in der Hauptsache den englisch- und deutschsprachigen Wikipedias.
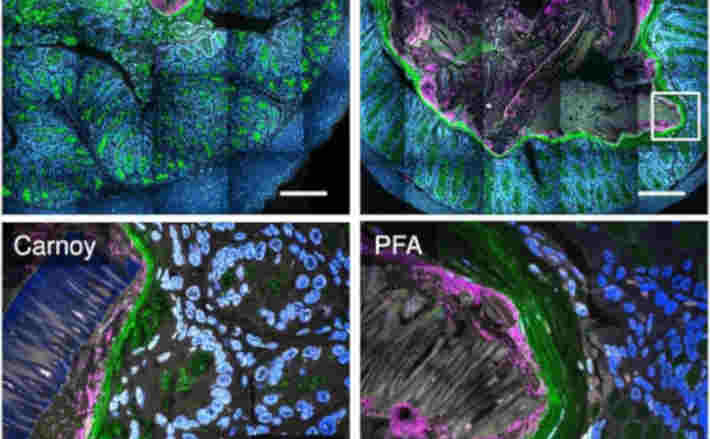
DAS TITELBILD
Das verwendete Foto stammt von von Hasegawa Y, Mark Welch JL, Rossetti BJ, Borisy GG CC BY 4.0 Es zeigt eine räumliche Struktur des intestinalen Bioms
Gleichzeitige Visualisierung von Schleim, reaktiven Mikroben mit FISH, Wirtsgewebe und aufgenommenen Lebensmitteln mit Carnoy-Fixierung und PFA-Fixierung.
Angrenzende 1 cm große Darmabschnitte wurden in Carnoy oder PFA fixiert, in Methacrylat eingebettet, geschnitten, parallel untersucht und mit identischen Bildeinstellungen aufgenommen.
Die im Mosaik gescannten Zusammenfassungsbilder (oben) zeigen die Position des Detailbildes (weißer Rahmen); Helligkeit und Kontrast wurden zur besseren Sichtbarkeit angepasst.
Die detaillierten Bilder (unten) zeigen ein großes Lebensmittelpartikel in der Nähe der Schleimhaut. Die Bilder wurden einer linearen Entmischung unterzogen und sind Projektionen von maximaler Intensität von bis zu 5 Ebenen.
DAPI färbt Nukleinsäuren, Weizenkeimagglutinin (WGA) färbt Schleim, und die Eub338-Sonde ist mit den meisten Bakterien hybridisiert. Skalenbalken =200 μm.
CC BY-SA 3.0